Intérêts de recherche : Ingénierie des protéines, biocatalyse, enzymologie, biologie structurale, modélisation moléculaire et dynamique des protéines.
La complexité de la catalyse enzymatique, qui consiste en une reconnaissance extrêmement spécifique des ligands suivie d'une transformation catalytique rapide, a traditionnellement été difficile à étudier. Les récents développements en matière de modélisation moléculaire des macromolécules et les puissantes méthodologies de biologie moléculaire offrent désormais de nouvelles approches pour améliorer notre compréhension des relations structure-fonction des enzymes. Notre programme de recherche combine ces méthodologies pour explorer les principes fondamentaux de la catalyse enzymatique. Les avantages d'une meilleure compréhension de la catalyse enzymatique sont nombreux : nous comprenons mieux les résistances d'origine enzymatique aux médicaments et nous modifions des enzymes pour des applications synthétiques. Actuellement, il y a un besoin urgent de développer des stratégies complémentaires afin de fournir plus de détails sur la nature des interactions enzyme-ligand pour des applications incluant la conception de médicaments et la biocatalyse. Pour améliorer notre capacité à modifier des enzymes à des fins de synthèse, nous obtenons des informations supplémentaires sur la plasticité des sites actifs des enzymes en distinguant les éléments indispensables à la fonction de ceux qui peuvent être modifiés. Nous développons et appliquons des méthodologies qui améliorent la vitesse, la puissance et les connaissances sur les différentes étapes impliquées dans l'ingénierie des enzymes.
Membres de l'équipe : Adem H.-Parisien, Megan-Faye Parker
Les transglutaminases (TGase) catalysent la formation de liaisons crosslink entre les protéines ou les peptides. Chez l'homme, la réticulation des protéines par les TGases est impliquée dans la coagulation du sang, l'endocytose, l'apoptose et peut également entraîner des troubles physiologiques tels que la formation de la cataracte et la maladie cœliaque. Il s'agit donc d'une cible pour la conception de médicaments. Dans le cadre d'une collaboration de longue date avec le professeur Jeffrey Keillor (Université d'Ottawa), nous avons généré un modèle pour la liaison des substrats peptidiques donneurs de petits acyles à la transglutaminase des tissus de mammifères en vue du développement de substrats et d'inhibiteurs modèles (Chica et al. (2004)), et avons exploré l'utilisation de substrats non natifs (Keillor et al.(2008)). En utilisant de nouveaux tests tels que ce test fluorométrique pour l'activité de transamidation (Gnaccarini et al. (2009)), nous avons découvert de puissants inhibiteurs cinnamoyles de la TGase des mammifères (Pardin et al.(2009); Pardin et al. (2008)).
Dans le but de développer son utilisation en tant que biocatalyseur, nous avons également démontré la propargylation de protéines spécifique au site en utilisant la transglutaminase des tissus de mammifères (Gnaccarini et al.(2012)). Nous nous sommes plus récemment tournés vers les transglutaminases microbiennes (MTG) comme alternative verte à la synthèse des amides. La MTG est largement utilisée dans l'industrie alimentaire et textile. La MTG est tolérante à une large gamme de conditions de réaction et est donc adaptée à un développement ultérieur en tant que biocatalyseur. Nous avons étudié une large gamme de substrats non naturels, de petites molécules, qui ont été choisis pour explorer la réactivité de l'enzyme et pour obtenir des produits synthétiques pertinents. Nos résultats indiquent qu'une variété d'amines fonctionne comme substrat accepteur, mais que des exigences plus strictes s'appliquent au substrat donneur. Nous avons démontré que la MTG peut être utilisée pour ajouter un alcyne ou un azide sur une chaîne peptidique, pour une réaction "Click" ultérieure (Gundersen et al. (2014)).

Membres de l'équipe : Abdelhadi Djaileb, Megan-Faye Parker, Daniela Quaglia
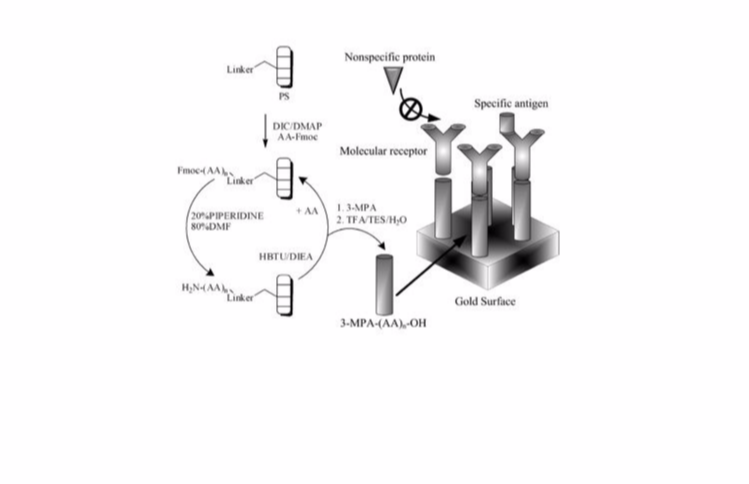
Les biomarqueurs sont des molécules spécifiques qui présentent une corrélation avec la progression des maladies ou avec la sensibilité d'une maladie à un traitement donné. Ce sont des indicateurs utiles pour suivre l'état d'avancement ou le traitement d'une maladie particulière. Nous développons des protocoles pour surveiller différents biomarqueurs liés au cancer en utilisant la résonance plasmonique de surface (SPR). La fixation d'une protéine d'intérêt particulière sur une surface en or permet la détection spécifique de l'interaction protéine-protéine ou la liaison d'une molécule cible d'intérêt en utilisant la SPR. Par exemple, la fixation d'un anticorps sur une surface en or permet la détection de son antigène spécifique dans des échantillons cliniques ou la fixation d'un antigène spécifique sur la surface peut permettre de surveiller le niveau d'anticorps, un indicateur utile de l'évolution du traitement de nombreuses maladies.
Nous développons des protocoles adaptables à un nouveau dispositif SPR portable conçu par le professeur Jean-François Masson. Nous avons développé et breveté différents types de monocouches de surface empêchant l'adsorption non spécifique qui permettent la détection SPR de molécules spécifiques en utilisant du sérum brut (Ratel et al. (2013); Aubé et al. (2013); Bolduc et al. (2011); Bolduc et al. (2010); Bolduc et al. (2009)). Nous utilisons cette approche pour surveiller le méthotrexate dans les échantillons cliniques de patients cancéreux (Zhao et al. (2012)). Nous avons maintenant développé différents nouveaux tests SPR pour la surveillance de l'évolution d'infections par le SRAS-CoV-2 (Djaileb et al. (2021); Hojjat Jodaylami et al. (2021); Djaileb et al. (2023)). Nous travaillons à l'amélioration de la sensibilité de la détection en optimisant les aspects physiques et biologiques du système SPR. Notre travail comprend l'ingénierie des protéines et la chimie des surfaces afin d'élaborer de nouveaux protocoles SPR pour améliorer l'accessibilité du diagnostic en surveillant la progression, le cours du traitement ou le dépistage de différentes maladies.
Membres de l'équipe : Claudèle Lemay-St-Denis, Stella Cellier-Goetghebeur, Maxime Saint-Aubin, Samy Faraj
L'utilisation croissante du triméthoprime comme antibiotique courant à usage médical et son application préventive dans l'élevage d'animaux terrestres et aquatiques ont augmenté sa prévalence dans l'environnement. Cela s'est accompagné d'une résistance accrue aux médicaments, notamment par l'émergence d'une dihydrofolate réductase de type B (DfrB) intrinsèquement résistante au TMP. La DfrB1, également connue sous le nom de dihydrofolate réductase R67, est la plus largement étudiée au sein de cette famille d'enzymes. Il est impératif de découvrir des inhibiteurs de cette nouvelle source de résistance aux antibiotiques, afin de maintenir l'efficacité du triméthoprime dans le traitement de maladies telles que la pneumonie, la gonorrhée et les infections adventices chez les patients atteints du SIDA.
Nous avons rapporté les premiers inhibiteurs spécifiques de cette cible médicamenteuse émergente, obtenus par conception à base de fragments (Bastien et al. (2012)). Nous avons développé une plateforme de criblage fonctionnel pour l'identification d'inhibiteurs sélectifs d'enzymes bactériennes de résistance au triméthoprime, les DfrB. La plateforme est basée sur la détermination semi-automatique de l'activité enzymatique en présence d'une variété de fragments, ou de composés simples similaires aux ligands natifs de l'enzyme, pour évaluer l'inhibition.
Nous avons observé qu'une variété de petits composés aromatiques offrent une inhibition millimolaire de la DfrB1, ce qui est cohérent avec la nature "primitive" proposée de son site de liaison relativement promiscuitaire (Schmitzer et al. (2004)). Par ces moyens, de petites molécules aromatiques de 150-250 g/mol (fragments) inhibant sélectivement la DfrB1 dans la gamme des faibles millimolaires ont été identifiées. Les molécules faiblement inhibitrices ont servi de base pour tester des composés de plus grande complexité, qui ont permis une augmentation de l'affinité du millimolaire au micromolaire. Des bis-benzimidazoles symétriques plus complexes et un bis-carboxyphényle ont permis une inhibition sélective de la cible dans la gamme des faibles micromolaires (Ki = 2-4 μM). Le docking moléculaire utilisant notre structure cristalline à haute résolution (Yachnin et al. (2011)) a permis de mieux comprendre le mode de liaison de ces inhibiteurs. De façon importante, nous avons criblé la dihydrofolate réductase humaine en parallèle avec le DfrB1 pour identifier les composés présentant les meilleures perspectives d'inhibition sélective. Nous avons ainsi identifié une nouvelle classe d'inhibiteurs sélectifs, symétriques et compétitifs de la DfrB1. Nous travaillons actuellement sur la prochaine génération d'inhibiteurs des DfrB.
Membres de l'équipe : Ali Fendri, Jonathan Besna, Douglas Fansher
Les monooxygénases P450 sont des enzymes contenant un groupement hème, qui sont utilisées en biocatalyse. Ces dernières années, l'utilisation des enzymes P450 comme biocatalyseurs a suscité un intérêt croissant en raison de leur capacité à effectuer des réactions hautement sélectives dans des conditions douces, réduisant ainsi l'impact environnemental des processus chimiques. La P450 BM3 de Bacillus megaterium hydroxyle divers substrats en utilisant le NADPH et l'oxygène moléculaire. Des mutations près de l'hème ont élargi sa gamme de substrats au-delà des acides gras. Les méthodes colorimétriques, telles que les essais basés sur les colonies bactériennes, accélèrent le dépistage de nouveaux substrats. L'élargissement de la gamme de substrats de la P450 BM3 pour les réactions industrielles pertinentes améliorerait l'efficacité et la durabilité en réduisant la dépendance à l'égard des catalyseurs chimiques agressifs. Cette enzyme polyvalente pourrait être utilisée dans les secteurs de la pharmacie, de l'agrochimie et de la chimie fine, donnant accès à de nouveaux échafaudages chimiques et à des stéréoisomères pour des produits de grande valeur.
L'adaptation de l'enzyme P450 BM3 à un système de réacteur à flux pourrait permettre de relever certains défis tels que la dépendance coûteuse aux cofacteurs, la réutilisation de l'enzyme et l'efficacité du découplage. La chimie en flux consiste à effectuer des réactions chimiques dans un réacteur à flux, tel qu'un dispositif capillaire ou micro-structuré, le temps de séjour déterminant alors le temps de réaction. Les réactifs sont d'abord pompés à travers un dispositif de mélange, puis conduits dans un réacteur à flux à température contrôlée. La technologie de la chimie en flux permet de mieux contrôler les paramètres de réaction, tels que le mélange, les concentrations, la température ou le temps de réaction. Cela signifie que nous pouvons tirer parti de réactions plus productives, plus sélectives et plus propres. Il s'agit d'une approche qui offre un meilleur contrôle de la réaction et réduit les réactions secondaires. Elle est donc radicalement différente de la chimie traditionnelle qui consiste à effectuer des réactions dans des flacons en verre ou dans des réacteurs à agitation continue.
À venir!
ADRESSE / ADDRESS
Université de Montréal
Département de Chimie
Campus MIL - Local B-6227
1375 Ave.Thérèse-Lavoie-Roux
Montreal, Qc, Canada, H2V 0B3
CONTACT
Phone: +1 (514) 343-2124
Fax: +1 (514) 343-7586
@ : joelle.pelletier@umontreal.ca